[导读] 译者:慧海拾穗
【摘要】
背景:霍奇金淋巴瘤(Hodgkin lymphoma, HL)主要由反应性、非肿瘤性细胞包围少见的肿瘤细胞,即所谓的Hodgkin and Reed-Sternberg(HRS)或淋巴细胞为主(lymphocyte dominant, LP)细胞组成。由于肿瘤细胞比较少,导致在很长一段时间内阻碍了肿瘤细胞基因组的分析,但最近发展起来的方法(特别是激光捕获显微解剖、流式细胞术/荧光激活细胞分类)促进了分子生物学的研究,阐明了“霍奇金淋巴瘤发生”的病理生理学原理。
方法:回顾近三十年来有关经典型和结节性淋巴细胞为主型HL(NLPHL)基因组图谱的相关文献,并总结其分子基础。结果:首先,HL恶性细胞通过各种基因改变引起的PDL1/2、B2M和MHC I、II类表达改变逃避免疫系统。其次,JAK/STAT信号通路中多个基因的普遍突变导致JAK/STAT信号被永久激活,从而促进肿瘤的生长。第三,NF-κB复合物和PI3K/AKT/mTOR轴的改变可以阻止肿瘤细胞凋亡。此外,Epstein-Barr病毒感染可同时激活JAK/STAT和NF-κB通路,类似地导致肿瘤细胞生存增强和凋亡逃避。最后,启动子高甲基化等表观遗传现象导致B系特异性基因、肿瘤抑制基因和免疫调节基因的下调。
结论:本文为HL的基因组学研究奠定了基础,为进一步深入研究其复杂的病理生理机制提供了理论依据。
【简介:大海捞针(Searching and Finding the Needle in the Haystack)】
霍奇金淋巴瘤在许多方面都是独特的。与其他许多恶性肿瘤不同的是,HL不仅逃脱了免疫控制,而且利用和招募了免疫系统来达到其目的,这导致了一个显著的、异质性的肿瘤微环境。事实上,在结节性淋巴细胞为主型HL(NLPHL)中的LP细胞,以及在经典型HL(cHL)中的霍奇金和Reed-Sternberg(HRS)细胞只占肿瘤的一小部分(通常小于1%)。因此,研究这些稀少的肿瘤细胞就像大海捞针,极大地阻碍了对各自肿瘤细胞基因组的分析,因为高通量分子研究方法需要较高的肿瘤细胞纯度和大量的输入DNA含量。然而,该领域的最新进展可能有助于HL基因组学的研究。
通过对细胞系的研究,人们对HL的遗传学有了更多的了解,这使得全外显子组测序等应用成为可能。然而,这些研究的有效性是有限的,因为它们不能代表HL复杂的肿瘤内异质性。特别是在cHL中,几乎所有的细胞系都来自于难治性和复发的病例,这绝不是绝大多数临床病程的反映。此外,目前只有一种NLPHL细胞株(DEV),一些真正的HL细胞株被误认为是这种细胞株。相比之下,从受累组织中通过激光捕获显微解剖(LCM)的肿瘤性LP和HRS细胞代表了一个更现实的方法,直到最近,在cHL的遗传学研究一直是最广泛使用的方法。在这项技术的帮助下,重现性基因组获得和缺失,以及单个候选基因的突变已经被识别出来。然而,LCM有重要的缺点,限制了它的使用。这种方法除了费时费力外,还涉及到高能激光辐射,可能会导致DNA损伤。此外,肿瘤细胞是从组织载玻片中收集的,载玻片中含有截断的细胞核,可能导致检测质量受损。Fromm等人介绍了一种使用流式细胞仪的替代方法,可以从新鲜或冷冻组织样本中快速分离和高纯度的HRS细胞。基于该方案,2015年首次对HRS细胞进行了全外显子组测序研究。2018年,我们的团队开发了一种基于荧光激活细胞分选(FACS)的新型富集技术,用于从福尔马林固定和石蜡包埋(FFPE)组织中分离HRS细胞。这包括DNA富集,全基因组扩增和68个基因的定制淋巴瘤组分析。尽管在没有潜在的激光辐射诱导DNA损伤的情况下富集数以万计的肿瘤细胞是可能的,但在FFPE中,保持足够的组织抗原性和对特定信号的细胞识别仍然是障碍。另一种有前途且优雅的方法是Spina等人报道的cHL患者循环肿瘤DNA(ctDNA)分析,Desch等人最近报道了儿童HL患者的循环肿瘤DNA分析【Genotyping circulating tumor DNA of pediatric Hodgkin lymphoma. Leukemia,2020,34:151-166.】。这种基于捕获的技术可以无创检测HRS细胞的突变,跟踪其在治疗过程中的克隆进化,并监测微小残留病灶。
在这些方法的帮助下,在过去的30年里,HL的细胞通路和基因改变的一般框架已经被阐明。1994年,通过显示具有严重突变的克隆重排免疫球蛋白重链和轻链(IG)基因,确定了HRS细胞是B细胞起源。在生理条件下,携带这种突变的B细胞会通过凋亡迅速被消灭。因此,为了破坏细胞凋亡诱导机制,HRS细胞操纵关键的细胞信号通路(主要是JAK/STAT和NF-κB),引入大量的救助通路(免疫逃逸和感染/ EBV的重新激活)以及基因启动子的高甲基化。因此,在HL中发现了多种被称为广泛性突变的基因。在下一节中,我们将根据受影响的途径讨论HL中已知的异常基因组(图1)。
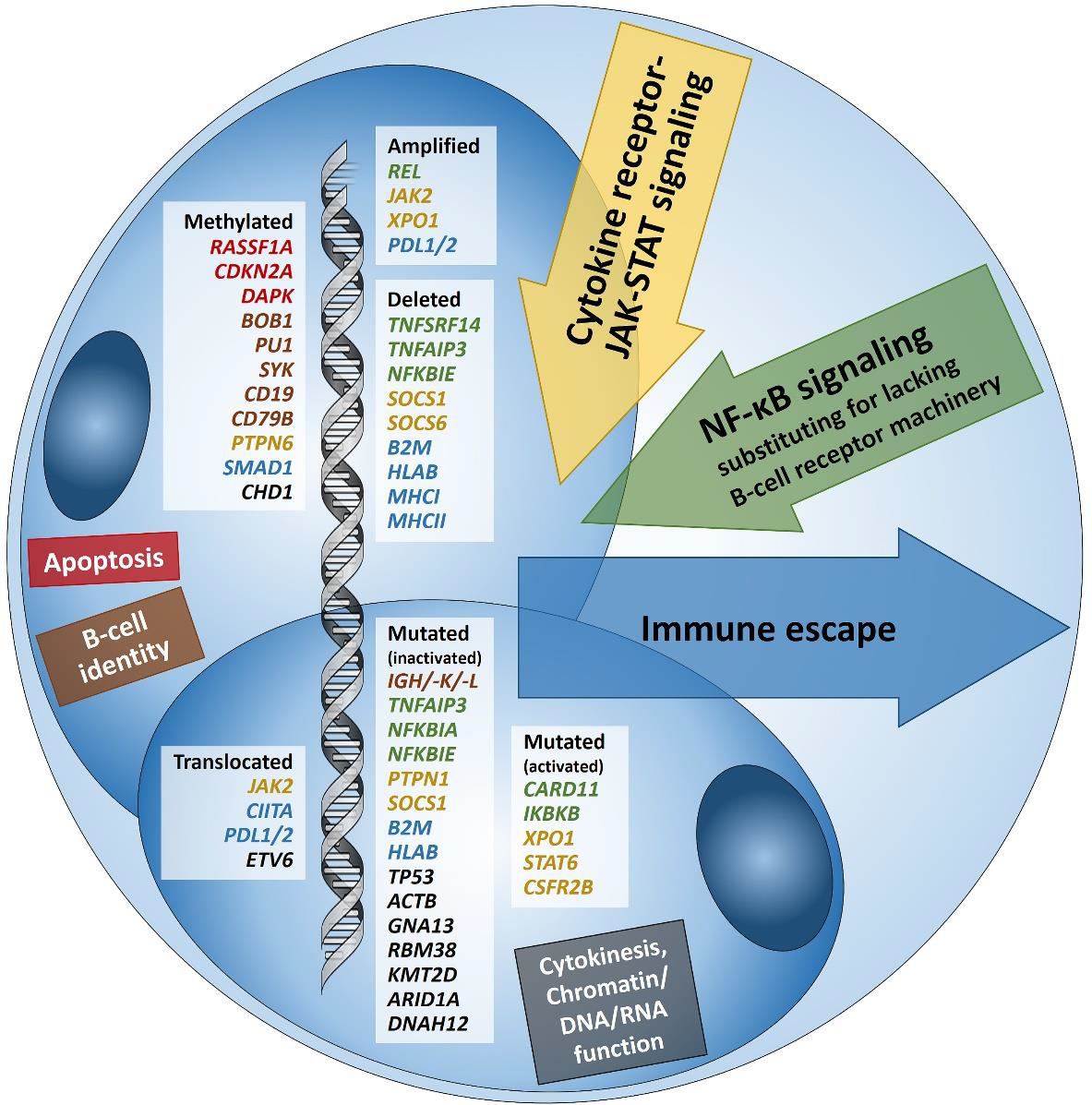
图1:经典型HL已知的异常基因相关通路
•••未完待续•••
点击下载原文文献












共0条评论