很像收银员利用机器扫描包装上的条形码来识别顾客在商店中购买的物品,科学家们利用显微镜和它们自身各种条形码来帮助辨别细胞的各个部分,或是疾病位点的分子类型。但他们的条形码只有极少数的“种类”,限制了科学家们在任何时间研究的细胞样品中对象的数量。
近日来自哈佛大学维斯生物工程研究所的研究人员制造了一种新型的条形码能够具有几乎无限的系列类型,相比以前有潜能使科学们在某个特定时间集合更多的重要信息。这种方法利用了DNA的天然自组装能力,在线报道在9月24日的《自然化学》(Nature Chemistry)杂志上。
领导这一研究的是华裔科学家印鹏(Peng Yin,音译),这位科学家主要研究方向是基于触发分子几何学的工程生物成像探针,曾荣获2010年美国NIH院长创新奖。
印鹏说:“我们希望这种新方法将为利用荧光显微镜研究复杂的生物学问题提供急需的分子工具。”
细胞成像的条形码新技术
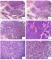
过去几十年里荧光显微镜一直是生物医学成像领域的一种绝技。简而言之,科学家们将荧光元件条形码连接到分子上,随后他们将分子附着到他们希望研究的细胞部分。照射样本触发每种条形码在特异的光波长发出如红色、蓝色或绿色的荧光,指明目的分子所在的位置。
然而,这种方法受到可获得颜色数量的限制——只有三或四种,有时候颜色还会变的模糊。这就是DNA条形码的魔力所在:各种颜色的小点以几何模式或荧光线性条码排列,且组合几乎是无限的,科学家们能够在一个样本中观察到的不同分子或细胞的数量显著增加,且颜色容易区分。
以下是它的工作原理:DNA折纸术(DNA origami)遵循着双螺旋的基本原子即分子碱基A(腺嘌呤)只结合T(胸腺嘧啶),而C(胞嘧啶)碱基只结合G(鸟嘌呤)。利用这些适当的“规定”,长链DNA在短链的帮助下按程序通过自身折叠进行自组装构建出预定的形式——很像将一张纸折叠构建出传统日本艺术中的各种图样。
对于这些结构上更加复杂的DNA纳米结构,研究人员能够随后将荧光分子附着到理想的位点,然后利用折纸技术用少数的荧光分子生成大量的条形码。由于它使得科学家们有可能比以往任何时候阐明更多的细胞结构,这可能会将许多添加到细胞成像的“工具箱”中。
“这些设计的DNA纳米结构固有的刚度是这种方法最大的优势:无需利用外力,它将荧光模式维持适当的位置。还大有希望利用这种方法在自然环境中研究细胞,”印鹏说。作为概念证明,研究小组证实他们其中的一个新条形码成功地附着到了酵母细胞表面。
还需要更多的研究来特别确定当每种荧光条形码一起混合在一种细胞样本中时会发生什么,这是在真实生物学和医学成像系统中的例行程序。但也有许多好消息作为起点,印鹏说相比当前的方法它的成本低、易操作且更为强大。
维斯研究所创会理事Don Ingber博士说:“我们正在快速推动利用折纸技术操作DNA分子的能力。它的潜力景观是巨大的,从帮助我们开发靶向药物传递机制到改善细胞和分子活动范围,我们能够利用最新的医学成像技术来观察疾病的位点。”











共0条评论